王明伟/Vogt/杨德华/水雯箐领衔中美科学家深度解析重要靶点PI3Kα的结构与功能
该论文运用纳米抗体(Nanobody)和化学交联(Chemical cross-linking mass spectrometry, XL-MS)相结合的方法,揭示了特异性纳米抗体通过引起构象变化影响磷脂酰肌醇3激酶α(Phosphoinositide 3 kinase alpha, PI3Kα)催化活性的分子机制和柔性结构域BH、cSH2及SH3的位置信息,为深入认识这个关键信号分子的结构与功能提供了新的实验依据。
PI3Kα是一种重要的脂质激酶,由催化亚基p110α和调节亚基p85α共同组成异二聚体,与细胞生长和增殖密切相关,其功能获得性突变是肿瘤形成的驱动因素1-2,因而成为治疗癌症的重要靶点。虽然PI3K抑制剂已被大量报道,但只有五种获准上市3-4。PI3Kα蛋白具有高度的灵活性,其结构解析困难重重5,全长复合物中迄今仍有三个柔性结构域(BH、cSH2和SH3)的相对位置尚不明了,严重阻碍了相关新药的创制。
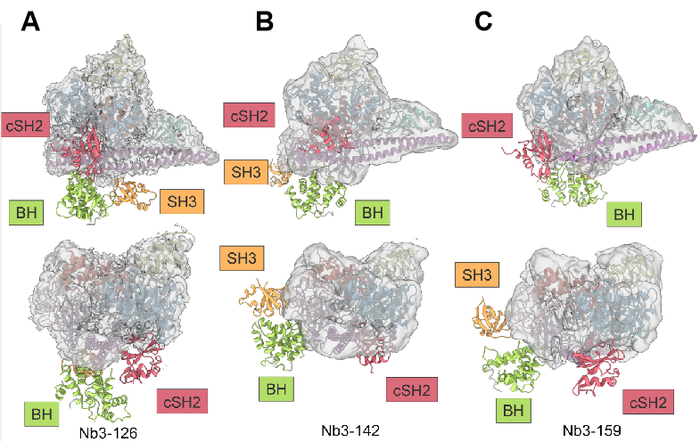
参加这项工作的科研人员首先对数十个PI3Kα特异性纳米抗体进行了筛选,从中选取3个高亲和力且稳定表达的抗体分子(Nb3-126、Nb3-142和Nb3-159),通过反复优化实验条件,获得了颗粒均一且性状稳定的全长PI3Kα分别与3种纳米抗体结合的蛋白复合物。借助中国科学院上海药物研究所300 kV冷冻电镜拍摄到的清晰图像和后继单颗粒三维重构,解析了整体分辨率分别为2.41、3.08和3.13 Å的立体结构。结果表明,对PI3Kα具有激活作用的Nb3-126使得催化核心具有高分辨率(图1A),但未能建模的p85α结构域密度则降低(图1B)。相反,抑制PI3Kα活性的Nb3-142使得催化核心的分辨率减弱,而未能建模结构域的密度却得以增强(图1C)。

图1. A,PI3Kα与Nb3-126复合物结构显示催化核心区域具有高分辨率。B,该复合物结构的局部分辨率图,显示未建模区域密度较低。C,PI3Kα与Nb3-142复合物结构显示其存在稳定的额外密度。
此外,在对酶活性无明显影响的Nb3-159与PI3Kα所形成复合物中的iSH2结构域发生显著移位(图2A)。三维可变性分析(3D variability analysis, 3DVA)发现了一种全新构象:iSH2和ABD位移超过10 Å并围绕其长轴旋转,nSH2结构域围绕p110α-E545-p85α-K379相互作用并略微旋转(图2B);随后的建模和均方根偏差值(root mean square deviation, RMSD)研究显示出PI3Kα与Nb3-159复合物构象的广泛变化(图2C)。

-
Burke JE, Perisic O, Masson GR, et al. Oncogenic mutations mimic and enhance dynamic events in the natural activation of phosphoinositide 3-kinase p110α (PIK3CA). Proceedings of the National Academy of Sciences USA, 2012, 109(38): 15259-15264. -
Fruman DA, Chiu H, Hopkins BD, et al. The PI3K pathway in human disease. Cell, 2017, 170(4): 605-635. -
Furet P, Guagnano V, Fairhurst RA, et al. Discovery of NVP-BYL719 a potent and selective phosphatidylinositol-3 kinase alpha inhibitor selected for clinical evaluation. Bioorganic & Medicinal Chemistry Letters, 2013, 23(13): 3741-3748. -
Krause G, Hassenrück F, Hallek M. Copanlisib for treatment of B-cell malignancies: the development of a PI3K inhibitor with considerable differences to idelalisib. Drug Design, Development and Therapy, 2018, 12: 2577. -
Vanhaesebroeck B, Burke JE, Madsen RR. Precision targeting of mutant PI3Kα in cancer by selective degradation. Cancer Discovery, 2022, 12(1): 20-22. -
Liu X, Yang S, Hart JR, et al. Cryo-EM structures of PI3Kα reveal conformational changes during inhibition and activation. Proceedings of the National Academy of Sciences USA, 2021, 118(45): e2109327118.
邵丽竹
何发
热点文章
-
制药分离技术在生物制药中的应用与探究
2025-12-12
-
中国药典2025年版“药品包装用塑料材料和容器指导原则”的框架体系与核心内容解析
2026-02-04
-
压片制粒生产各过程关键因素之考量
2025-12-22
-
药品共线生产中的清洁验证与风险管控
2025-12-25
-
生物制品分段委托生产情形下的持有人质量管理体系关注点探讨
2025-12-16
-
制药行业未来趋势洞察:数智化、个性化、可持续与全球化重塑产业格局
2025-12-10
-
底喷微丸包衣工艺参数对包衣效果的影响分析
2025-12-04
-
基于CFD仿真技术的灌装机充氮装置设计优化
本文以某制药产线的灌装机设备为研究对象,采用计算流体动力学(CFD)仿真技术对充氮装置的充氮性能进行分析,并结合分析结果对氮幕结构进行了优化设计。随后,针对优化方案进行性能仿真验证,结果显示优化后的顶空残氧量降低至0.252%。为了进一步验证优化方案的实际效果,将优化方案应用于实际产线进行性能测试,测得的顶空残氧量为0.68%,这一结果满足了小于1%的要求,表明其充氮保护性能已达到国际先进水平。
作者:王志刚、刘依宽、刘佳鑫
-
药品密封性检测 :用户需求与优化
-
可控冻融系统在生物原液上的应用
-
人用疫苗生产数字化转型
-
药包材生产质量管理的进阶策略
-
药厂洁净区域风量和压差的控制策略













评论
加载更多