Nature Review | 靶向RNA小分子的研发策略、优化、十年展望
现有的RNA靶向小分子使用一系列机制,包括通过与细胞蛋白作为分子胶实现定向剪接,抑制不可药物蛋白的翻译和非编码RNA中的功能结构失活。最近发表在Nature Reviews Drug Discovery上的综述“Targeting RNA structures with small molecules”概述了识别、验证和优化靶向功能转录组的小分子研发策略及其未来展望。
1.1 确定RNA结构
1.2 评估RNA结构预测的准确性
1.3 定义转录组中的功能RNA结构
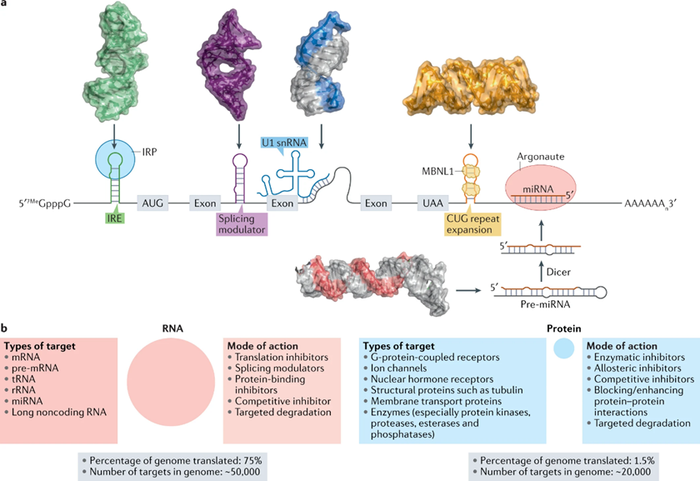
图1:人类RNA调节关键的生物过程,其功能由其结构驱动
1.4 影响小分子靶向RNA选择性的因素
1.5 结合RNA的小分子之间的共性
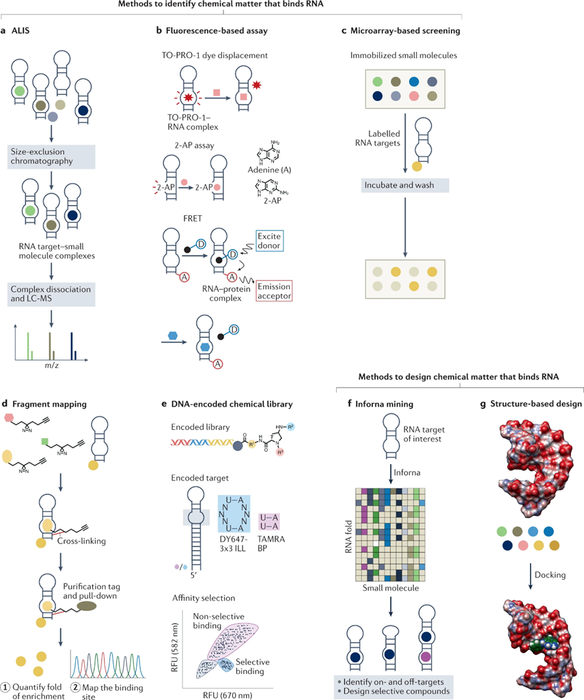
2.1 以RNA为中心的方法
2.2 设计小分子RNA结合剂
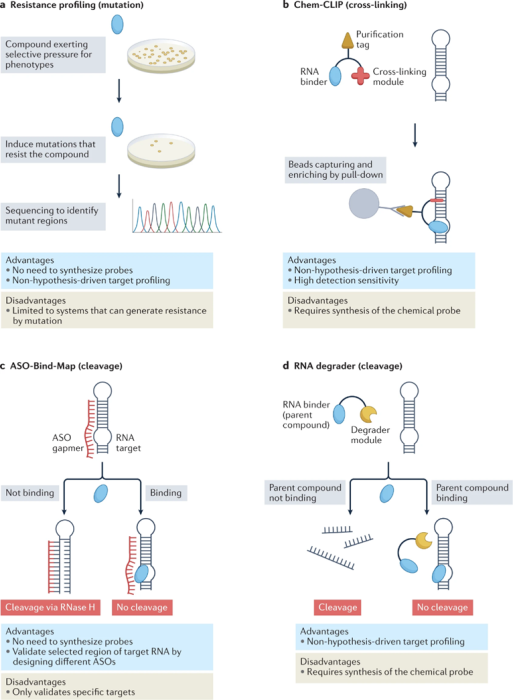
3.1 耐药性分析
3.2 共价键的形成来测量小分子与靶RNA的直接接触
3.3 靶RNA切割以评估结合占用率
3.4 量化选择性
4.1 传统的药物化学方法。
4.2 结构导向方法。
4.3 模块化装配方法。
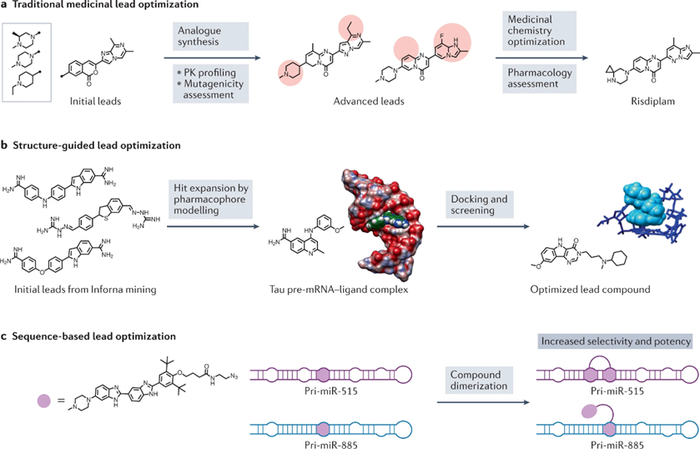
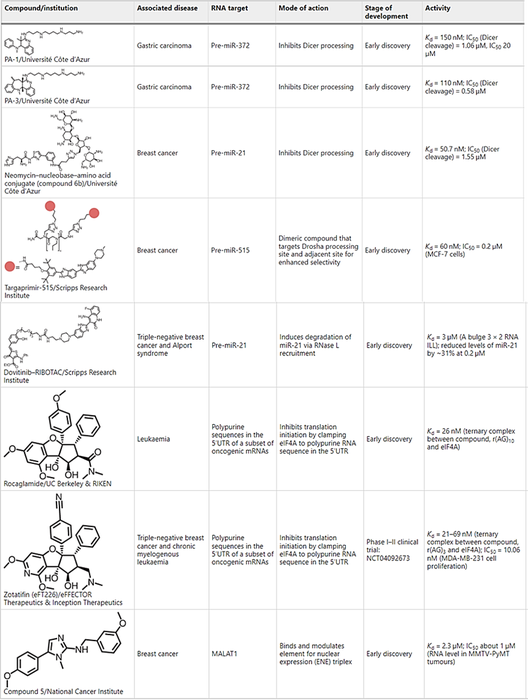
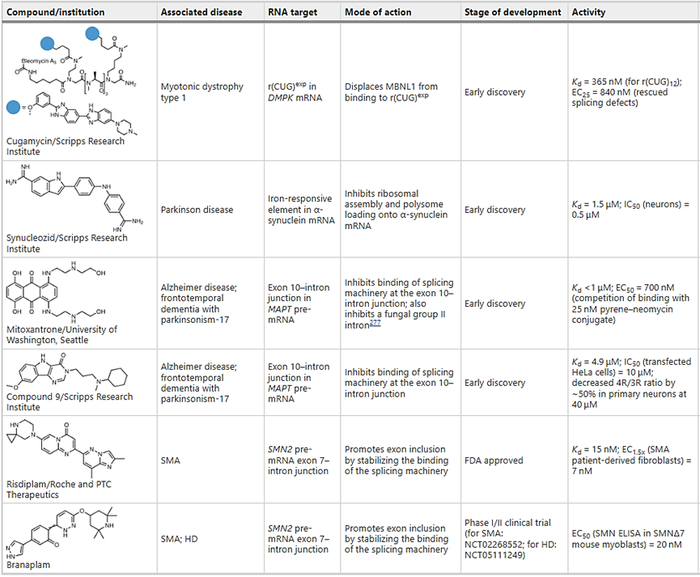
5.1 靶向功能性RNA结构
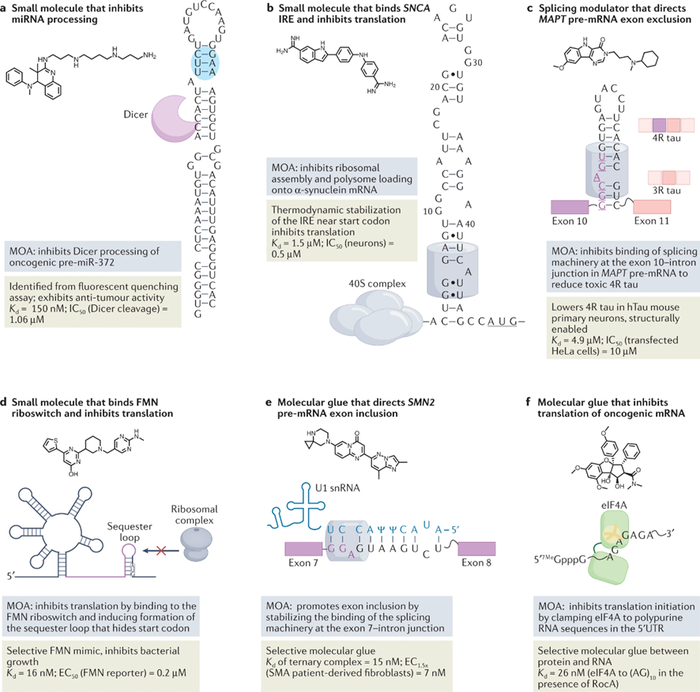
5.2 靶向RNA结构进行降解
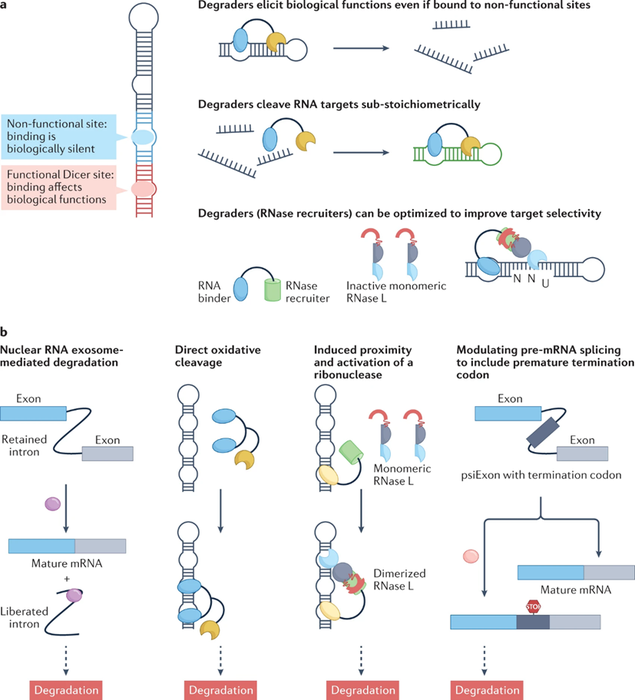
5.3 靶向RNA相关的通路

来源:BiG生物创新社
邵丽竹
何发
热点文章
-
制药分离技术在生物制药中的应用与探究
2025-12-12
-
中国药典2025年版“药品包装用塑料材料和容器指导原则”的框架体系与核心内容解析
2026-02-04
-
压片制粒生产各过程关键因素之考量
2025-12-22
-
药品共线生产中的清洁验证与风险管控
2025-12-25
-
生物制品分段委托生产情形下的持有人质量管理体系关注点探讨
2025-12-16
-
制药行业未来趋势洞察:数智化、个性化、可持续与全球化重塑产业格局
2025-12-10
-
底喷微丸包衣工艺参数对包衣效果的影响分析
2025-12-04
-
基于CFD仿真技术的灌装机充氮装置设计优化
本文以某制药产线的灌装机设备为研究对象,采用计算流体动力学(CFD)仿真技术对充氮装置的充氮性能进行分析,并结合分析结果对氮幕结构进行了优化设计。随后,针对优化方案进行性能仿真验证,结果显示优化后的顶空残氧量降低至0.252%。为了进一步验证优化方案的实际效果,将优化方案应用于实际产线进行性能测试,测得的顶空残氧量为0.68%,这一结果满足了小于1%的要求,表明其充氮保护性能已达到国际先进水平。
作者:王志刚、刘依宽、刘佳鑫
-
药品密封性检测 :用户需求与优化
-
可控冻融系统在生物原液上的应用
-
人用疫苗生产数字化转型
-
药包材生产质量管理的进阶策略
-
药厂洁净区域风量和压差的控制策略













评论
加载更多